ВВЕДЕНИЕ
Реконструкция генных сетей с помощью методов биоинформатики позволяет определять молекулярные и функциональные взаимодействия между генами, ассоциированными с изучаемым заболеванием, определять центральные, узловые гены в сети с целью более эффективного лекарственного воздействия. Компьютерное исследование ассоциированных с заболеванием генов, анализ их взаимодействий расширяют применение интернет-технологий в медицине [1, 2].
Мы рассматриваем анализ генов синдрома Ангельмана – расстройства развития нервной системы, характеризующегося серьезными трудностями в обучении, атаксией, судорожным расстройством, едва заметными дисморфическими чертами лица и изменениями поведения. У большинства детей наблюдается задержка в развитии, не развивается речь [3].
Пациенты с синдромом Ангельмана имеют характерный поведенческий фенотип с резкими движениями, частым и иногда неуместным смехом, расстройством сна. Больные имеют тонкие черты лица, широкий улыбающийся рот, выдающийся подбородок и глубоко посаженные глаза. Такой фенотип вызван различными генетическими аномалиями, включающими область хромосомы 15q11-13, которая подвержена геномному импринтингу. Генетические механизмы обнаруживаются у 85-90% пациентов с клиническим фенотипом, и все они связаны с экспрессией гена UBE3A (ubiquitin-protein ligase E3A), распложенного на данном участке хромосомы 15 [4, 5]. UBE3A демонстрирует тканеспецифичный импринтинг, экспрессируясь исключительно от материнской аллели в головном мозге. В то же время с развитием технологий секвенирования, получением новых геномных данных появляется новая информация о генах и их продуктах, которые могут быть функционально связаны с синдромом Ангельмана. Актуальным является исследование синдрома Ангельмана с помощью современных средств биоинформатики.
Компьютерная реконструкция взаимодействия макромолекул (генов и белков), ассоциированных с исследуемым заболеванием, задает генную сеть – матрицу взаимодействий генов и графическую структуру. Определение ключевых генов заболевания, полученных с помощью анализа структуры генной сети, важно для дальнейшего определения наиболее значимых генов в структуре сети с целью дальнейшего подбора вариантов фармакологических воздействий для лечения [6]. Сама методика использования компьютерных средств для анализа сетевых взаимодействий генов, ассоциированных с наследственными заболеваниями, представляет большой научный интерес. Подобные работы по реконструкции генных сетей с помощью онлайн инструментов биоинформатики проводились нами для других комплексных заболеваний, в частности болезни Паркинсона и шизофрении [7, 8]. В настоящее время компьютерные средства биоинформатики развиваются все более активно, включая онлайнплатформы, средства статистической обработки и визуализации генных сетей, интегрированные с базами данных [6, 8]. Представлены программные конвейеры обработки списков генов, включающие наборы взаимно дополняющих онлайн-инструментов [9].
Решение задачи анализа генов синдрома Ангельмана началось с построения актуального списка ассоциированных генов по существующим базам данных генетической информации, верификации генов по представленности в различных базах. Далее с помощью инструментов биоинформатики выполнена функциональная аннотация, определение категорий генных онтологий для списка генов, выполнена реконструкция генной сети, построена визуализация графической структуры. В нашей работе представлена методика реконструкции генной сети на примере синдрома Ангельмана. Выделены узловые элементы сети, кластеры генов, показаны сетевые взаимодействия с другими заболеваниями.
МАТЕРИАЛЫ И МЕТОДЫ
Составление списка генов, связанных с наследственной предрасположенностью к синдрому Ангельмана, выполнено с помощью интернет-ресурса OMIM (Online Mendelian Inheritance in Man) [10] (https://omim.org/) и GeneCards (GeneCards®: The Human Gene Database) (https://www.genecards.org/) [11]. Эти онлайн ресурсы интегрируют экспериментальную информацию по генам человека и их фенотипическим проявлениям из других баз данных, аннотацию генов NCBI GenBank, функциональную аннотацию белков из базы UniProt (https://uniport.org), информацию по нуклеотидным полиморфизмам из dbSNP (Single Nucleotide Polymorphism database) [12].
По классическому определению синдрома Ангельмана к заболеванию можно отнести лишь ген UBE3A. Интерес представляет расширение списка генов ассоциированных генов, меняющих экспрессию у пациентов, с доказанной связью с фенотипом заболевания. Запрос «Angelman syndrome» к базе данных секвенирования SRA (Short Reads Archive) (https://www.ncbi.nlm.nih.gov/sra) [13] показывает экспериментальные работы данных секвенирования, относящиеся к заболеванию – 338 наборов данных, из которых лишь половина относится к данным по человеку, остальные выполнены на модельных животных. Для определения генов заболевания после перерасчета данных можно выделить дифференциально экспрессирующиеся гены, как ассоциированные с заболеванием. Был выбран путь поиска ассоциированных генов по запросам к базам данным, интегрирующим уже известную экспериментальную информацию.
Ресурсы OMIM [10] и GeneCards [11] были использованы для анализа генов менделевского наследования у человека, поиск проводился по запросу «Angelman syndrome». По нашему запросу (Search: «angelman syndromе» (Search in: Entries with: Genemap; Retrieve: gene map)) база данных OMIM выдает 56 результатов, которые включают 139 официальных символов гена. База данных GeneCards выдает 1,116 результатов.
Реконструкцию генной сети взаимодействий генов синдрома Ангельмана проводили с помощью двух ресурсов STRING-DB [14] и GeneMANIA [15]. Найдено разное количество генов в базе, используемой конкретным ресурсом.
Ресурс GeneMANIA (Multiple Association Network Integration Algorithm) ориентирован на анализ ассоциаций, представленных в научной литературе, с возможностью интерактивного перестроения (отрисовки) графа генной сети онлайн (https://genemania.org). Онлайн-ресурс и база данных STRING (Search Tool for the Retrieval of Interacting Genes) (https://string-db.org/) изначально был ориентирован на анализ белок-белковых взаимодействий, обладает гибким функционалом, возможностями перерисовки, редактирования структуры сети онлайн, статистических оценок связности сети, выполнения операций кластеризации (выделения связных элементов сети).
Анализ категорий генных онтологий выполнялся с помощью ресурса DAVID (Database for Annotation, Visualization and Integrated Discovery) (https://david.ncifcrf.gov/summary.jsp, новый сайт – https://davidbioinformatics.nih.gov/) [16]. Для анализа онтологий по основным группам – биологические процессы, молекулярные функции, клеточные компартменты – использовался ресурс PANTHER (Protein Analysis Through Evolutionary Relationships, http://pantherdb.org) [17].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Используя ресурсы OMIM и GeneCards [11], по запросу angelman syndrome был получен актуальный список 240 наименований генов. Рассмотрим функциональную аннотацию генов синдрома Ангельмана, интегрируя источники информации с помощью ресурса GeneCards [11].
Онлайн-платформа GeneCards дает оценки релевантности запроса (ассоциированности термина с искомым заболеванием) в вариантах GIFtS (GeneCards Inferred Functionality Scores) по представленности в базах данных [18], и Score («счет» – число упоминаний заданного термина в аннотации каждого конкретного гена в базах данных). Рассмотрим наиболее релевантные гены (приоритезированный список), по GeneCards из более чем 1000 генов (по запросу Аngelman syndrome) (табл. 1).
Из таблицы видно, что первые места по ассоциированности с синдромом Ангельмана занимают Убиквитин Протеинлигаза E3A (UBE3A), как и отмечается в литературе [4, 5], Циклинзависимая Киназа, Подобная 5 (CDKL5), и МетилCpG-связывающий белок 2 (MECP2). Большая часть списка – белок-кодирующие гены.
Интерес представляет описание общих молекулярных функций для генов из полного списка. Далее был выполнен расчет обогащенных категорий и анализ генных онтологий для полного списка генов синдрома Ангельмана с помощью ресурса DAVID. Дополнительно ресурс, ресурс DAVID позволяет рассчитывать перепредставленность терминов по другим базам данных, например по базе данных метаболических путей KEGG (Kyoto Encyclopedia of Genes and Genomes) (представлено группой категорий KEGG_PATHWAY) (https://www.kegg.jp/).
Таблица 1. Гены, ассоциированные с синдромом Ангельмана по данным CeneCards
Table 1. Cene, associated with Angelman syndrome by CeneCards annotation
| № | Официальное имя гена Official gene name |
Описание гена Gene description |
Категория Category |
GIFtS* | Счет ** Score ** |
|---|---|---|---|---|---|
| 1 | UBE3A | Убиквитин-протеинлигаза E3A Ubiquitin Protein Ligase E3A |
Белок-кодирущий ген Protein Coding |
57 | 135,76 |
| 2 | CDKL5 | Циклинзависимая киназа, подобная 5 Cyclin Dependent Kinase Like 5 |
Белок-кодирущий ген Protein Coding |
52 | 113,38 |
| 3 | MECP2 | Белок, связывающий метил-CpG 2 Methyl-CpG Binding Protein 2 |
Белок-кодирущий ген Protein Coding |
55 | 111,98 |
| 4 | TP53 | Опухолевый белок P53 Tumor Protein P53 |
Белок-кодирущий ген Protein Coding |
62 | 107,04 |
| 5 | FBN1 | Фибриллин 1 Fibrillin 1 |
Белок-кодирущий ген Protein Coding |
56 | 105,08 |
| 6 | SNHG14 | Ген-хозяин малой ядрышковой РНК 14 Small Nucleolar RNA Host Gene 14 |
Ген длинной некодируюшей РНК (lncRNA) RNA Gene (lncRNA) |
24 | 101,50 |
| 7 | PTCH1 | Патчированный 1 Patched 1 |
Белок-кодирущий ген Protein Coding |
60 | 95,58 |
| 8 | KMT2D | Лизинметилтрансфераза 2D Lysine Methyltransferase 2D |
Белок-кодирущий ген Protein Coding |
53 | 73,87 |
| 9 | CDH1 | Кадхерин 1 Cadherin 1 |
Белок-кодирущий ген Protein Coding |
58 | 71,61 |
| 10 | SNRPN | Полипептид малого ядерного рибонуклеопротеина N Small Nuclear Ribonucleoprotein Polypeptide N |
Белок-кодирущий ген Protein Coding |
53 | 60,17 |
| 11 | NLRP3 | Пириновый домен семейства NLR, содержащий 3 NLR Family Pyrin Domain Containing 3 |
Белок-кодирущий ген Protein Coding |
57 | 58,88 |
| 12 | PEX1 | Фактор биогенеза пероксисом 1 Peroxisomal Biogenesis Factor 1 |
Белок-кодирущий ген Protein Coding |
53 | 57,03 |
| 13 | AR | Рецептор андрогена Androgen Receptor |
Белок-кодирущий ген Protein Coding |
60 | 56,23 |
| 14 | CDKN2A | Ингибитор циклинзависимой киназы 2A Cyclin Dependent Kinase Inhibitor 2A |
Белок-кодирущий ген Protein Coding |
60 | 54,55 |
| 15 | BBS4 | Синдром Барде-Бидля 4 Bardet-Biedl Syndrome 4 |
Белок-кодирущий ген Protein Coding |
50 | 54,35 |
| 16 | CDKN1C | Ингибитор циклин-зависимой киназы 1C Cyclin Dependent Kinase Inhibitor 1C |
Белок-кодирущий ген Protein Coding |
53 | 53,16 |
| 17 | NSD1 | Белок, связывающий набор доменов ядерного рецептора 1 Nuclear Receptor Binding SET Domain Protein 1 |
Белок-кодирущий ген Protein Coding |
53 | 50,19 |
| 18 | KIF7 | Член семейства кинезинов 7 Kinesin Family Member 7 |
Белок-кодирущий ген Protein Coding |
48 | 48,09 |
| 19 | POLGARF | Альтернативная рамка считывания POLG POLG Alternative Reading Frame |
Белок-кодирущий ген Protein Coding |
17 | 47,30 |
| 20 | SLC9A6 | Семейство растворимых носителей 9, член A6 Solute Carrier Family 9 Member A6 |
Белок-кодирущий ген Protein Coding |
54 | 46,61 |
Примечание: * счет (оценка) функциональной аннотации гена GIFtS (GeneCards Inferred Functionality Scores) по представленности в базах данных, следуя [18];
** счет релевантности оценивает соответствие поисковому запросу в базе данных по синдрому Ангельмана (Angelman syndrome).
Note: * score (estimate) of gene functionalityа GIFtS (GeneCards Inferred Functionality Scores) is counted following method described in [18];
** score of relevance to the search term in the databases (for term Angelman syndrome).
Таблица 2 содержит Категории генных онтологий с указанием группы данных в базе DAVID (Биологические процессы – GOTERM_BP_DIRECT, Клеточные компоненты – GOTERM_CC_DIRECT, Молекулярные функции – GOTERM_MF_DIRECT, Пути KEGG – KEGG_PATHWAY); термины онтологий, которые обогащены в данном наборе генов; процент генов в выборке; уровень значимости для перепредставленности онтологий в данном набоек P-Value и корректированное P* (коррекция Бонферрони на множественность гипотез). Из таблицы 2 видно, что наиболее значимыми группами онтологий для генов синдрома Ангельмана являются регуляция транскрипции, хроматин, развитие синапсов, работа гормонов (оригинальные термины positive regulation of transcription from RNA polymerase II promoter, chromatin, postsynapse, GABA-A receptor complex, hormone activity, Endocrine resistance). Действительно, данное заболевание связано с развитием, функцией гормонов, и вовлеченность таких категорий онтологий обоснована.
Таблица 2. Категории генных онтологий для генов, ассоциированных с синдромом Ангельмана, рассчитанные с помощью ресyрса DAYID
Table 2. Cene ontology categories for the genes associated with Angelman Syndrome, calculated using the DAYID resource
| Категории онтологий Group of ontologies |
Термины онтологий Ontology term |
% генов % genes |
Знач. P P-value |
P* |
|---|---|---|---|---|
| Биологические процессы GOTERM_BP_DIRECT |
Реакция на эстрадиол Response to estradiol |
5,8 | 7,0E-12 | 1,7E-8 |
| Биологические процессы GOTERM_BP_DIRECT |
Положительная регуляция транскрипции с промотора РНК-полимеразы II Positive regulation of transcription from RNA polymerase II promoter |
17,5 | 2,7E-10 | 6,4E-7 |
| Клеточные компоненты GOTERM_CC_DIRECT |
Хроматин Chromatin |
15,0 | 4,1E-9 | 1,7E-6 |
| Клеточные компоненты GOTERM_CC_DIRECT |
Постсинапс Postsynapse | 5,4 | 2,6E-8 | 1,1E-5 |
| Клеточные компоненты GOTERM_CC_DIRECT |
ГАМК-рецепторный комплекс GABA-A receptor complex |
2,9 | 3,1E-8 | 1,3E-5 |
| Молекулярные функции GOTERM_MF_DIRECT |
Гормональная активность Hormone activity |
5,0 | 2,8E-8 | 1,5E-5 |
| Пути KEGG KEGG_PATHWAY |
Эндокринная резистентность Endocrine resistance |
5,4 | 6,2E-8 | 1,6E-5 |
| Молекулярные функции GOTERM_MF_DIRECT |
Активность ГАМК-рецепторов GABA-A receptor activity |
2,9 | 3,5E-8 | 1,8E-5 |
| Молекулярные функции GOTERM_MF_DIRECT |
ДНК-связывающая DNA binding |
16,2 | 3,8E-8 | 2,0E-5 |
| Биологические процессы GOTERM_BP_DIRECT |
Cинаптическая передача, ГАМКергическая Synaptic transmission, GABAergic |
3,3 | 1,1E-8 | 2,7E-5 |
| Пути KEGG KEGG_PATHWAY |
Взаимодействие нейроактивных лиганд-рецепторов Neuroactive ligand-receptor interaction |
9,6 | 1,3E-7 | 3,3E-5 |
| Пути KEGG KEGG_PATHWAY |
Старение клеток Cellular senescence |
6,2 | 2,5E-7 | 6,6E-5 |
| Рецептор андрогена Androgen Receptor |
Активность ГАМК-зависимых хлорид-ионных каналов GABA-gated chloride ion channel activity |
2,5 | 1,6E-7 | 8,6E-5 |
| Пути KEGG KEGG_PATHWAY |
Рак поджелудочной железы Pancreatic cancer |
4,6 | 4,1E-7 | 1,1E-4 |
| Пути KEGG KEGG_PATHWAY |
Пути развития рака Pathways in cancer |
11,2 | 4,5E-7 | 1,2E-4 |
| Биологические процессы GOTERM_BP_DIRECT |
Положительная регуляция каскада МАРК Positive regulation of MAPK cascade |
5,8 | 5,3E-8 | 1,3E-4 |
| Биологические процессы GOTERM_BP_DIRECT |
Положительная регуляция экспрессии генов Positive regulation of gene expression |
9,6 | 6,2E-8 | 1,5E-4 |
| Клеточные компоненты GOTERM_CC_DIRECT |
Синапс Synapse | 8,8 | 4,4E-7 | 1,8E-4 |
| Клеточные компоненты GOTERM_CC_DIRECT |
Проекция нейронов Neuron projection |
6,7 | 1,0E-6 | 4,2E-4 |
| Молекулярные функции GOTERM_MF_DIRECT |
Активность внеклеточных ионных каналов, управляемых лигандами Extracellular ligand-gated ion channel activity |
2,9 | 8,4E-7 | 4,4E-4 |
| Клеточные компоненты GOTERM_CC_DIRECT |
Комплекс хлоридных каналов Chloride channel complex |
3,3 | 1,5E-6 | 6,3E-4 |
| Молекулярные функции GOTERM_MF_DIRECT |
Связывание протеинкиназы Protein kinase binding |
8,3 | 1,2E-6 | 6,4E-4 |
| Молекулярные функции GOTERM_MF_DIRECT |
Связывание хроматина Chromatin binding |
8,3 | 1,4E-6 | 7,3E-4 |
| Клеточные компоненты GOTERM_CC_DIRECT |
ГАМКергический синапс GABAergic synapse |
3,8 | 2,0E-6 | 8,3E-4 |
| Пути KEGG KEGG_PATHWAY |
Глиома Glioma |
4,2 | 3,5E-6 | 9,2E-4 |
Примечание к таблице: P* – корректированное по Бонферрони значение уровня значимости p
Table Note: P* – corrected by Bonferroni P-value
Из таблицы видна обогащенность набора генов синдрома Ангельмана категориями онкологических заболеваний, например глиомы, что может быть связано с нарушениями работы клеток мозга.
Для дальнейшего уточнения категорий онтологий на независимой базе данных использовался ресурс PANTHER (Protein ANalysis THrough Evolutionary Relationships) (http://pantherdb.org/). Данные PANTHER ассоциированы с полной базой онтологий генов человека ресурса Gene Ontology (https://geneontology.org/ ). Из набора исходных генов часть идентификаторов были не распознаны системой. С помощью PANTHER построена таблица онтологий (табл. 3) для категорий биологических процессов. Для получения наиболее информативных результатов значения P были ограничены с учетом коррекции Бонферрони. Таблица содержит три стандартных раздела: онтологии для биологических процессов, молекулярных функций и клеточных компонент (компартментов), обогащения числа генов в наборе в разы (параметр Fold Change) по сравнению с ожидаемым, и корректированное значение P (вероятность получить такое число генов по случайным причинам).
Таблица 3. Категории генных онтологий для генов синдрома Ангельмана, рассчитанные с помощью ресyрса PANTHER
Table 3. Cene ontology categories for genes associated with Angelman syndrome calculated by PANTHER tool
| Категории генных онтологий для биологических процессов Gene Ontology Categories for Biological Processes |
FC* | P* |
|---|---|---|
| Регуляция процесса развития Regulation of developmental process |
3,43 | 1,94E-16 |
| Процесс развития Developmental process |
2,3 | 2,06E-16 |
| Системы развития System development |
2,72 | 4,98E-15 |
| Развитие многоклеточного организма Multicellular organism development |
2,57 | 9,56E-15 |
| Реакция на эндогенный стимул Response to endogenous stimulus |
4,5 | 1,59E-14 |
| Процессы многоклеточного организма Multicellular organismal process |
2,07 | 2,26E-14 |
| Развитие анатомических структур Anatomical structure development |
2,31 | 5,53E-14 |
| Регуляция биологического качества Regulation of biological quality |
2,64 | 1,06E-13 |
| Категории генных онтологий для молекулярных функций Gene Ontology Categories for Molecular Functions |
FC* | P* |
| Гормональная активность Hormone activity |
3,43 | 2,24-E7 |
| Активность анионных каналов, управляемых лигандами Ligand-gated anion channel activity |
2,3 | 1,87E-6 |
| ГАМК-управляемая активность хлорид-ионного канала GABA-gated chloride ion channel activity |
2,72 | 1,46E-5 |
| Сигналы связывающих рецепторов Signaling receptor binding |
2,57 | 2,28E-5 |
| Белковый связывающий комплекс Protein-containing complex binding |
4,5 | 8,13E-5 |
| Активность ГАМК-рецептора GABA-A receptor activity |
2,07 | 9,2E-5 |
| Категории клеточных компартментов Cellular compartment categories |
FC* | P* |
| Синапсы Synapse |
3,81 | 3,49E-10 |
| Ципоплазматические везикулы Cytoplasmic vesicles |
2,78 | 2,0E-9 |
| Внутриклеточная везикула Intracellular vesicle |
2,78 | 2,09E-09 |
| Везикулы Vesicle |
2,19 | 5,0E-8 |
| Белковый комплекс Protein-containing complex |
1,88 | 1,17E-7 |
| Мембранные органеллы Membrane-bounded organelle |
1,36 | 2,16E-7 |
| Внутриклеточные мембранные органеллы Intracellular membrane-bounded organelle |
1,39 | 7,68E-7 |
Примечание: FC* (fold change) – превышение наблюдаемого числа генов в наборе к ожидаемому (по общей доле генов данной категории онтологий в геноме)
Note: FC* (fold change) – ratio of observed number of genes in the sample to the number expected by chance
Приведенные в таблице 3 оценки обогащенности онтологий показывают, что наиболее значимыми для генов синдрома Ангельмана по биологическим процессам являются категории регуляции процесса развития, регуляции на эндогенный стимул и развитие анатомических структур. По молекулярным функциям наиболее значимыми являются категории гормональной активности, активность анионных каналов, связывания рецепторов, белков, а также активность ГАМК-рецепторов. Для клеточных компартментов наиболее значимые категории в проявлении синдрома Ангельмана – синапсы, везикулы и другие категории, связанные с нервной тканью. Данные таблиц 2 и 3, построенных с помощью разных программных инструментов DAVID и PANTHER для исследуемых генов синдрома Ангельмана, подтверждают общие категории генных онтологий – это процессы развития организма, гормональная активность, компоненты нейрона. Таким образом, можно дать функциональное описание данного заболевания как болезни роста, нервной системы с нарушениями работы мозга.
Далее для реконструкции и визуализации генной сети ассоциированных с синдром Ангельмана генов использовались онлайн-инструменты GeneMANIA и STRING-DB (https://string-db.org/). Структура сети по GeneMANIA (http://genemania.org/search/homo-sapiens/UBE3A/CDKL5/MECP2/TP53/FBN1/SNHG14/PTCH1/KMT2D/CDH1/SNRPN/NLRP3/PEX1/AR/CDKN2A/BBS4/CDKN1 C/NSD1/KIF7/POLGARF/SLC9A6//) представлена на рисунке 1.
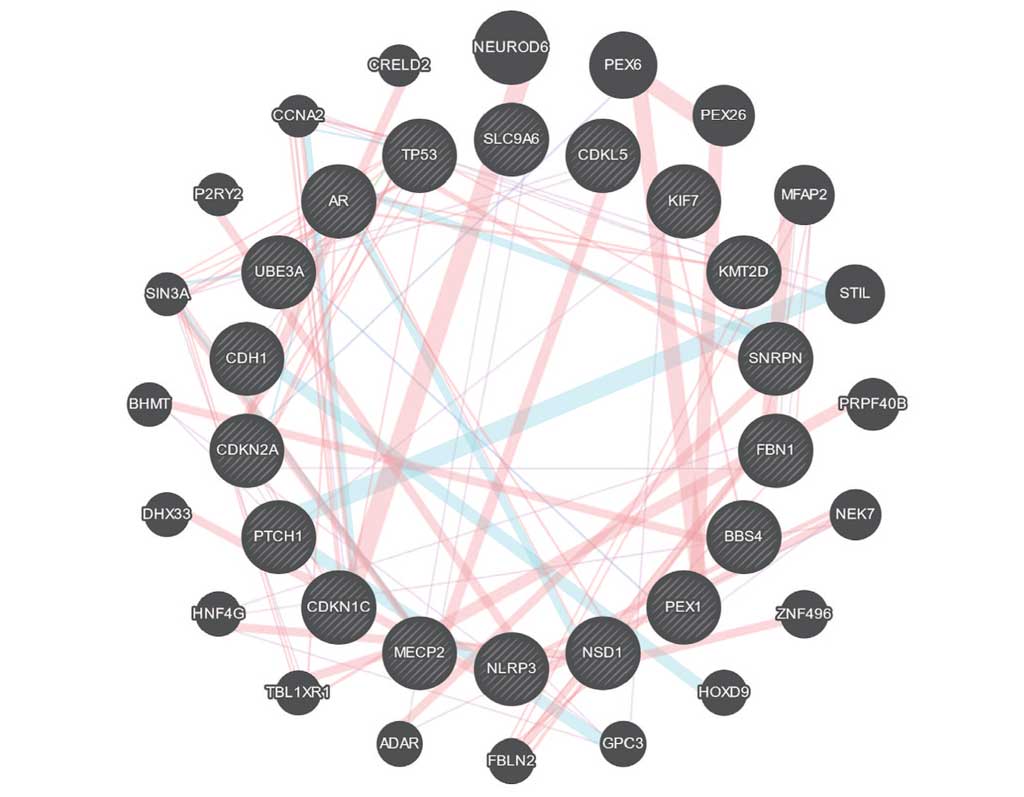
Рис. 1. Генная сеть генов синдрома Ангельмана, реконструированная с помощью ресурса GeneMANIA (http://genemania.org/)
Fig. 1. Gene network of genes associated with Angelman syndrome reconstructed by GeneMANIA (http://genemania.org/)
На рисунке 1 показана генная сеть взаимодействий для 20 генов синдрома Ангельмана (табл. 1), реконструированная с помощью онлайнинструмента GeneMANIA (http://genemania.org/). Сеть включает взаимодействия, автоматически построенные по литературным данным и собранные на данном открытом ресурсе. Заданные по списку гены показаны на рисунке большими черными кругами со штриховкой, контактирующие с ними гены представлены кругами меньшего размера. Расположение по окружности является одним из параметров визуализации системы GeneMANIA.
В построенной сети (рис. 1) находятся гены (продукты генов – белки), имеющие большое число связей с другими элементами – UBE3A, MECP2, SNHG14. При этом видно, что не все узлы связаны друг с другом.
Гены в реконструированной сети имеют взаимодействия разных типов, установленные как экспериментально (физические взаимодействия – Physical interaction), так и по косвенным данным (совместная экспрессия, совместная локализация в геноме Co-expression, Co-localization) (рис. 2).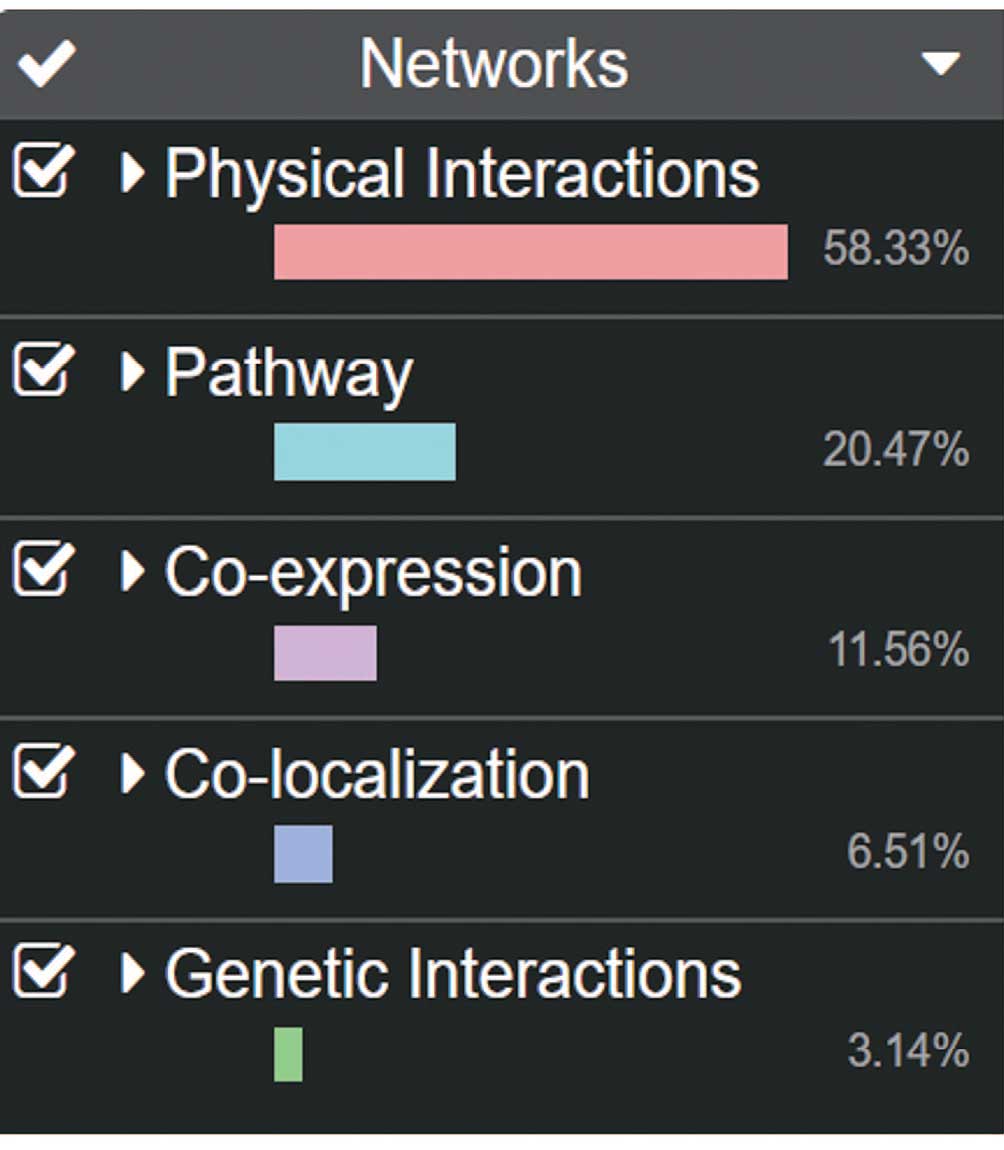
Рис. 2. Параметры взаимодействий для генов синдрома Ангельмана, представленные в инструменте GeneMANIA
Fig. 2. Interaction parameters for Angelman syndrome genes presented in the GeneMANIA tool
Основные типы взаимодействий (выделено цветом), использованные при построении сети на основе литературных источников – белок-белковые контакты (физические взаимодействия), совместная экспрессия, в меньшей степени – генетические взаимодействия.
Рисунок 2 показывает большее участие физических взаимодействий между макромолекулами в данной сети.
Далее мы использовали ресурс STRING-DB для реконструкции генной сети по заданному списку генов, расширив его до 240 имен генов (официальных символов – official gene symbol). Использовались параметры по умолчанию (белок-белковые контакты, экспериментальные данные, совместная локализация генов, аналогично используемым в инструменте GeneMANIA). Из-за большого числа связей разных типов сеть представляет собой сложный рисунок. Мы использовали ограничение визуализации в STRING-DB только на экспериментально доказанные взаимодействия.
Рисунок 3 представляет результат реконструкции генной сети синдрома Ангельмана с помощью STRING-DB с учетом только экспериментально доказанных взаимодействий.
Статистика структуры сети показала, что сеть имеет неслучайно большое число связей, хотя есть и не связанные гены. Связи соответствуют определенным взаимодействиям – физическим, генетическим, регуляторным, как и в системе GeneMANIA (рис. 1). Статистические оценки структуры сети генов синдрома Ангельмана в STRING-DB (https://string-db.org/) показывают высокую связность (значимость <1,0E-16). Средняя степень связности узла сети – 3.21, коэффициент кластеризации – 0.357. Общее число узлов сети (распознано имен генов) – 205, число связей между ними – 329 (при ожидаемом по случайным причинам – 166). На рисунке можно выделить несколько кластеров сети, самый большой из которых включает гены UBE3A, GABRB3, ATP10A, SNRPN. Кластеры сети показаны на рисунке 4 (кластеризация выполнена по методу k-средних (k-means clustering), инструмент в программе STRING-DB).
Большой кластер (показан красным цветом) занимает центральную позицию полученной сети и более детально представляет большую структуру сети, построенную по тем же генам на рисунке 3. Кластер связан с наибольшим числом других объектов.
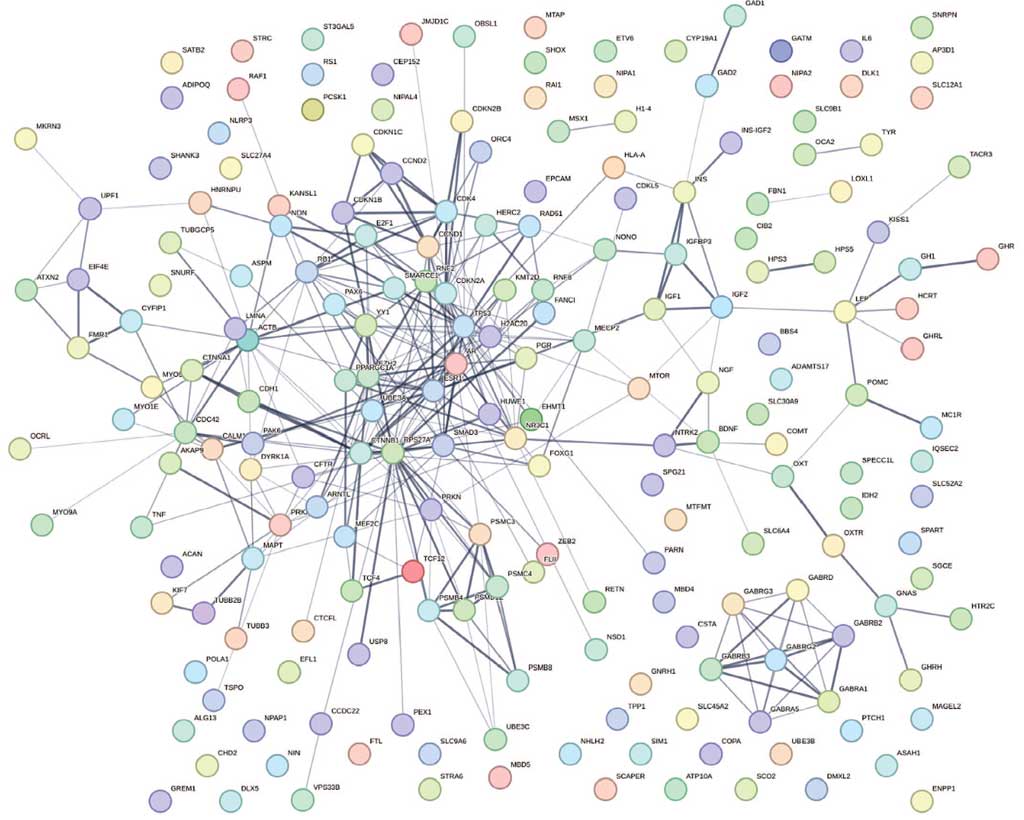
Рис. 3. Реконструкция сети взаимодействий генов синдрома Ангельмана с помощью STRING-DB (https://string-db.org/)
Fig. 3. Reconstruction of the network of interactions of Angelman syndrome genes using STRING-DB (https://string-db.org/)
На рисунке 4 видно присутствие в сети кластеров генов меньшего размера, не связанных непосредственно с основным кластером, включающим UBE3A.
Ген UBE3A кодирует убиквитин-белковую лигазу Е3, входящую в систему деградации белка убиквитина [3, 4]. Этот импринтированный ген экспрессируется в мозге и биаллельно экспрессируется в других тканях. Унаследованная от матери делеция этого гена вызывает синдром Ангельмана, характеризующийся выраженной моторной и интеллектуальной задержкой, атаксией, гипотонией, эпилепсией, отсутствием речи и характерными чертами лица. Белок взаимодействует с белком Е6 вируса папилломы человека типов 16 и 18, что приводит к убиквитинированию и протеолизу опухолевого белка р53.
Мутации в гене CDKL5 были связаны с синдромом Х-сцепленного инфантильного спазма (синдром Веста) и синдромом Ретта [19]. Также отмечается, что мутации CDKL5 шире, чем сообщалось ранее. Они являются важной причиной инфантильных спазмов и ранних эпилептических припадков у пациенток женского пола, а также более поздних трудноизлечимых судорожных расстройств [19].
Взаимосвязь между заболеваниями рассматривалась с помощью ресурса MalaCards [20], интегрирующего данные по генетическим заболеваниям и расстройствам. Реконструкция сети заболеваний возможна по общим признакам, связывающим каждую пару заболеваний, таким как гены, метаболиты, симптомы, общие лекарственные средства, упоминание в литературе. Рассмотрим аннотацию самого заболевания на ресурсе MalaCards (https://www.malacards.org/card/ angelman_syndrome), входящем в платформу GeneCards (рис. 5).
На рисунке 5 видна связь синдрома Ангельмана с хромосомными нарушениями, синдромом Ретта, с синдромом Прадера-Вилли, что подтверждается наличием общих генов и общих симптомов.
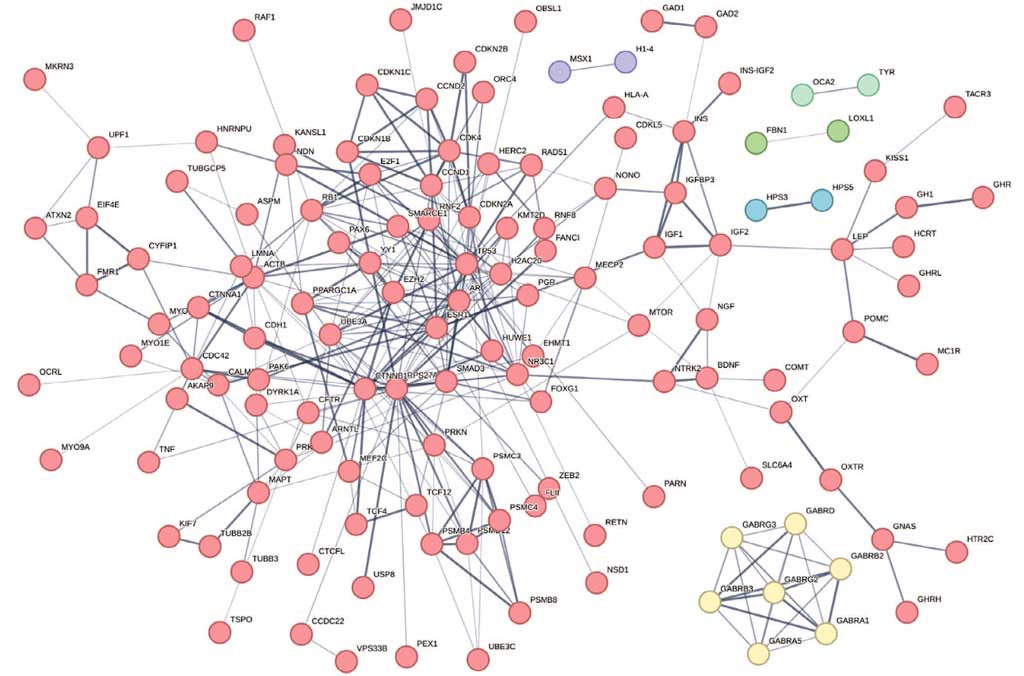
Рис. 4. Кластеры сети взаимодействий генов синдрома Ангельмана. Кластеризация выполнена в STRING-DB (https://string-db.org/)
Fig. 4. Clusters of the network of interactions of the genes of Angelman syndrome. Clustering is performed in STRING-DB (https://string-db.org/)
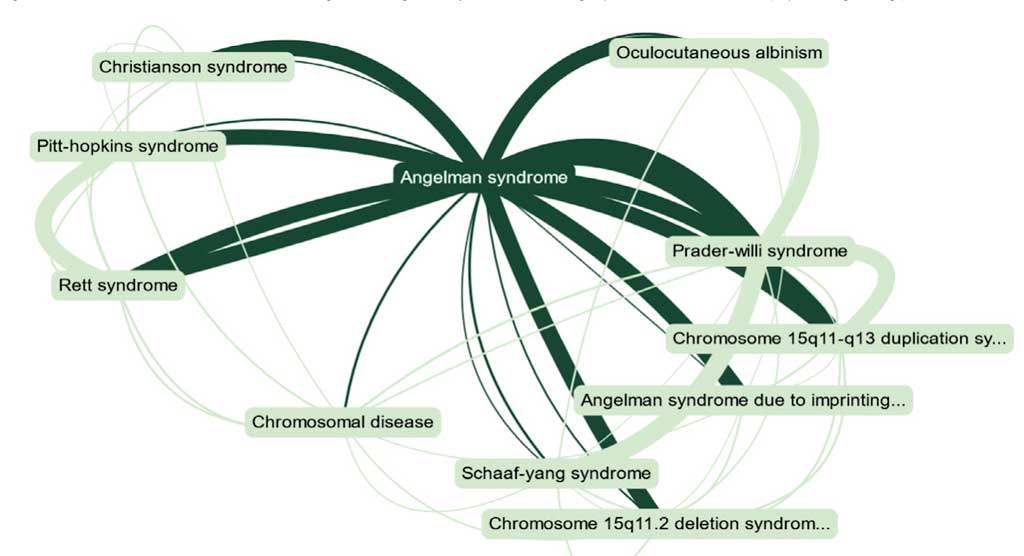
Рис. 5. Связанные заболевания для синдрома Ангельмана по MalaCards (https://www.malacards.org/card/angelman_syndrome)
Fig. 5. Related diseases for Angelman syndrome by MalaCards (https://www.malacards.org/card/angelman_syndrome)
Ресурс MalaCards представляет свой набор генов, так называемые «элитные» гены, описанные в литературе и ассоциированные с данным заболеванием (https://www.malacards.org/card/angelman_syndrome). Список представлен в таблице 4.
Список генов в Таблице 4 отличается от других списков, сохраняя, тем не менее, самые важные гены, такие как UBE3A, а также показывает число публикаций в PubMed, описывающих связь гена с заболеванием – синдромом Ангельмана. Интересно отметить, что наибольшее число связей в генной сети соответствует месту гена синдрома Ангельмана в приоретизированном списке как по Таблице 1 (построенной с помощью ресурса GeneCards), так и по таблице 4 (MalaCards).
Связь между заболеваемостью синдромом Ангельмана и локусом, содержащим гены, наиболее интересна для изучения, поскольку мутации в хромосомной области человека 15q11-q13 имеют гены, импринтированные по материнской линии (т. е. экспрессированные по отцовской линии), гены, импринтированные по отцовской линии (т. е. экспрессированные по материнской линии), и биаллельно экспрессированные гены [21].
Генетические исследования человека показали, что синдром Ангельмана вызван 4 молекулярными механизмами: de novo материнскими делециями хромосомы 15q11-q13 (70-80%); внутригенными мутациями в унаследованном от матери UBE3A в пределах хромосомы 15q11-q13 (10-20%); отцовской унипарентальной дизомией для хромосомы 15q11-q13 (3-5%); дефектами импринтинга в хромосоме 15q11-q13, изменяющими экспрессию унаследованного от матери UBE3A (3-5%). Существует вариабельность для каждого молекулярного класса синдрома Ангельмана: пациенты с делецией имеют более тяжелый фенотип, а те, у кого наблюдаются дефекты импринтинга, менее тяжелый фенотип [22, 23]. Известны случаи заболевания расстройствами аутического спектра и синдромом Ангельмана, вызванные общей хромосомной делецией [22].
ЗАКЛЮЧЕНИЕ
Реконструкция генных сетей, основанная на анализе данных генов, вызывающих синдром Ангельмана, приводит к выявлению сетевых структур, функционально связанных с генами риска заболевания. Обнаружение функциональных связей будет иметь жизненно важное значение для разработки эффективных подходов к лечению. Учитывая совпадение симптомов с другими нарушениями развития нервной системы, ожидается, что эти данные будут широко применяться.
Исследование связанных заболеваний (сети заболеваний), в частности, с помощью MalaCards позволяет определить лекарственные средства, уже применяемые для лечения других заболеваний. Представленное использование онлайн инструментов биоинформатики создает методический задел для анализа генов редких наследственных заболеваний, расширяя применение для других заболеваний [7, 8]. Дальнейшее исследование синдрома Ангельмана требует объединения клинических и биоинформационных исследований для построения более точных моделей.
Таблица 4. Наиболее важные («элитные») гены синдрома Ангельмана по данным MalaCards (https://www.malacards.org)
Table 4. The most important («elite») genes of Angelman syndrome according to MalaCards (https://www.malacards.org)
| № | Имя гена Gene Name |
Описание гена Gene description |
Тип гена Gene type |
Число публикаций Number of publications |
|---|---|---|---|---|
| 1 | UBE3A | Убиквитин-протеинлигаза E3A Ubiquitin Protein Ligase E3A |
Белок-кодирущий ген Protein Coding |
100 |
| 2 | SNHG14 | Ген-хозяин малой ядрышковой РНК 14 Small Nucleolar RNA Host Gene 14 |
Ген РНК RNA Gene |
41 |
| 3 | MECP2 | Белок, связывающий метил-CpG 2 Methyl-CpG Binding Protein 2 |
Белок-кодирущий ген Protein Coding |
20 |
| 4 | CDKL5 | Циклинзависимая киназа, подобная 5 Cyclin Dependent Kinase Like 5 |
Белок-кодирущий ген Protein Coding |
1 |
| 5 | GABRG3 | Субъединица рецептора Гамма-аминомасляной кислоты типа А Гамма 3 Gamma-Aminobutyric Acid Type A Receptor Subunit Gamma3 |
Белок-кодирущий ген Protein Coding |
|
| 6 | TPP1 | Трипептидилпептидаза 1 Tripeptidyl Peptidase 1 |
Белок-кодирущий ген Protein Coding |





































